readme:
运行顺序
我们拟分三批抓取论文信息
- 首先抓取详细信息,包括文章的
题目,第一作者,单位,期刊,关键词 - 其次抓取简要信息,包括文章的
题目,全部作者,期刊名称,发表时间,被引次数,下载次数 - 最后抓取文章的
题目和摘要
这里要说明的是,我分三次抓取的最主要原因是,知网详细界面和简略界面所显示的内容有所差异,为了全方位获取文章的信息,我们分批次抓取,最后可根据文章题目按需横向合并(merge)
我们首先手动操作,感受下检索过程,便于明确爬虫各个步骤的操作。
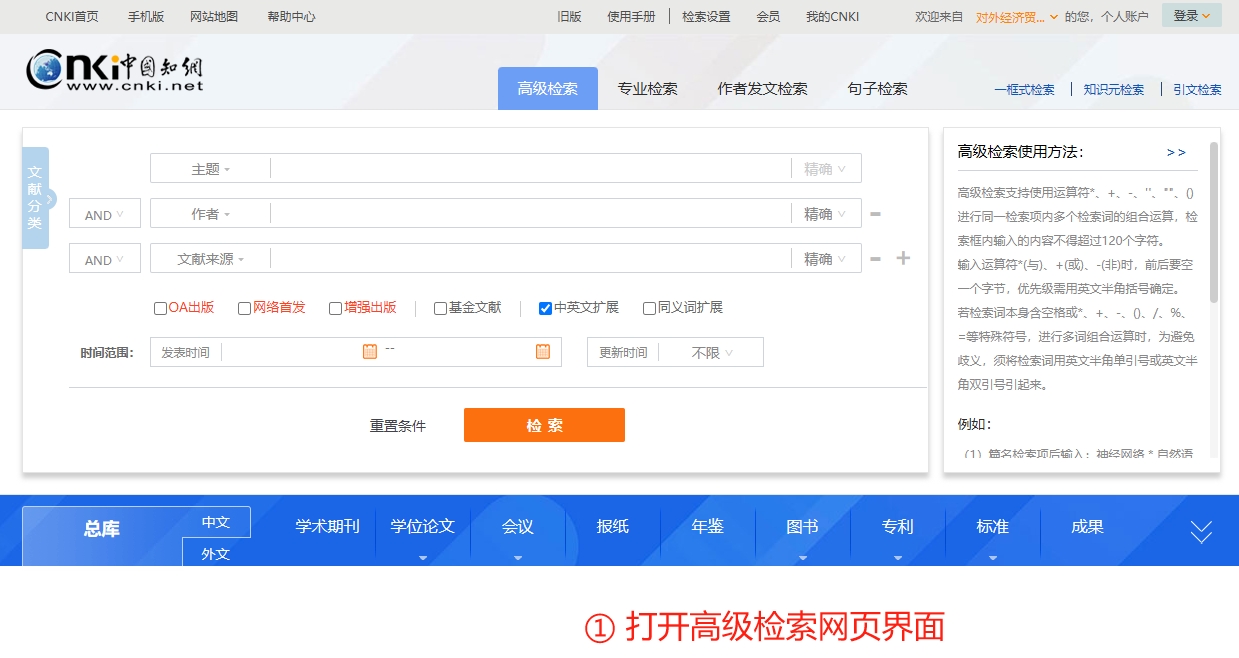
首先第一步是打开知网高级检索网页,这一步应该没有任何困难
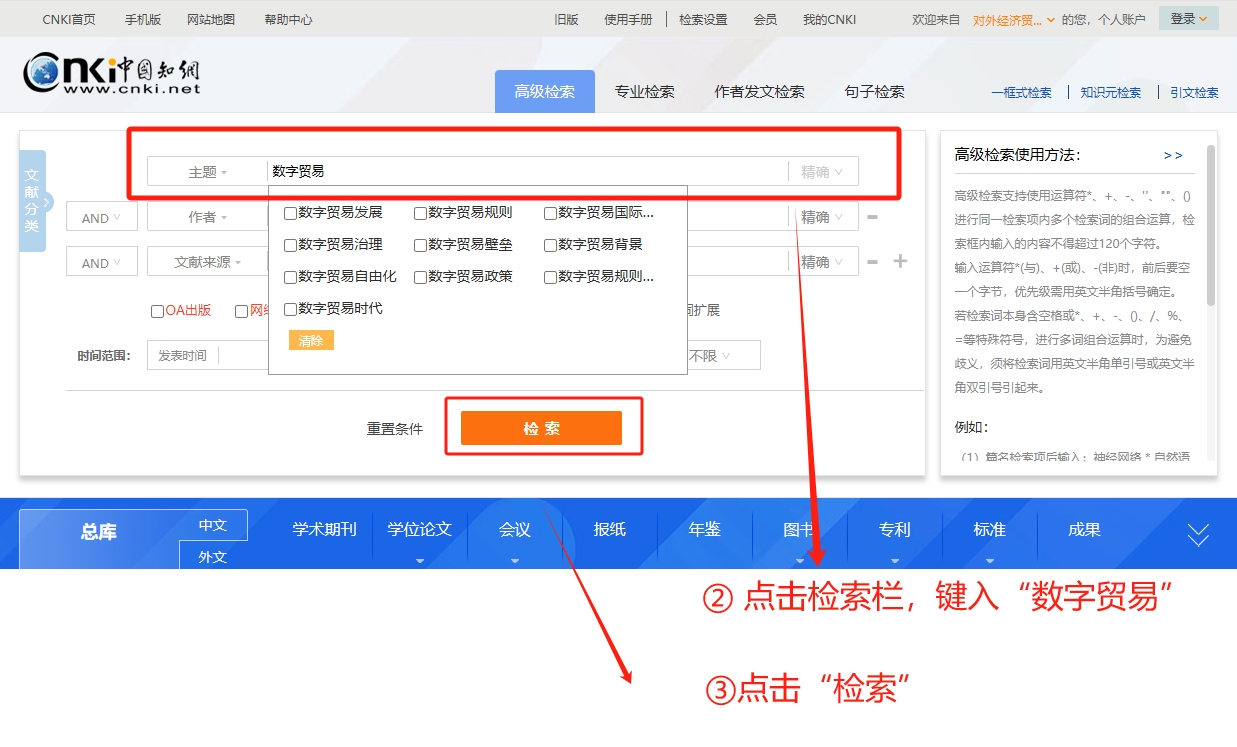
然后,我们点击主题栏,再键入“数字贸易”,完成后点击“检索”
如下图所示
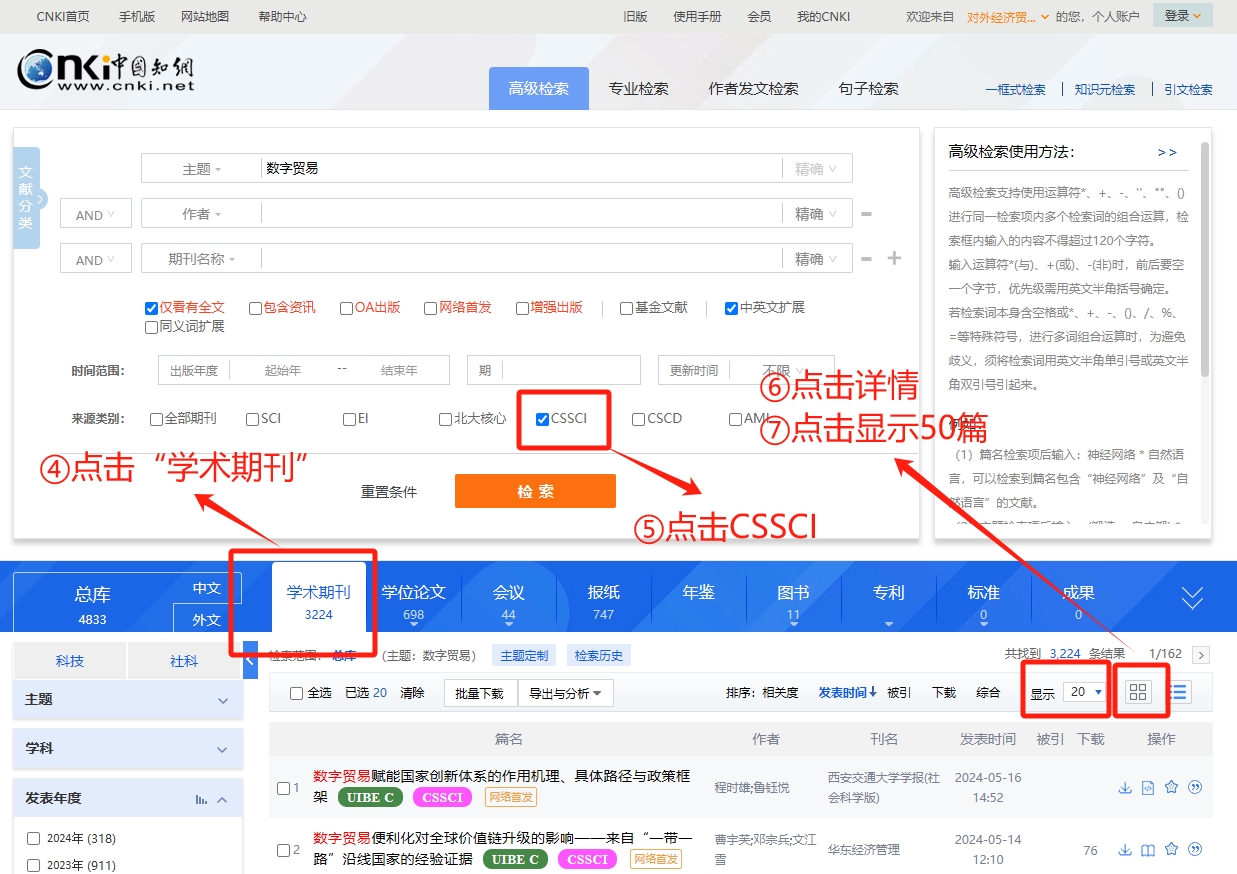
在点击完检索后,会跳出如下界面。此时,我们要爬取的是学术期刊,因此先点击④所示的“学术期刊”。在点击完成后,期刊类型(CSSCI、CSCD、AMI)就会自动显示,此时如果我们要筛选南大核心期刊的话,还需要勾选CSSCI前的方框。
最后,为了显示论文的详细信息,我们要点击如⑥所示的详细信息。
为了减少翻页,提高单一页面的阅读容量,我们还可以把“每页20篇”下拉选项框改为“每页50篇”
至此,我们就人为模拟了文献的检索过程。
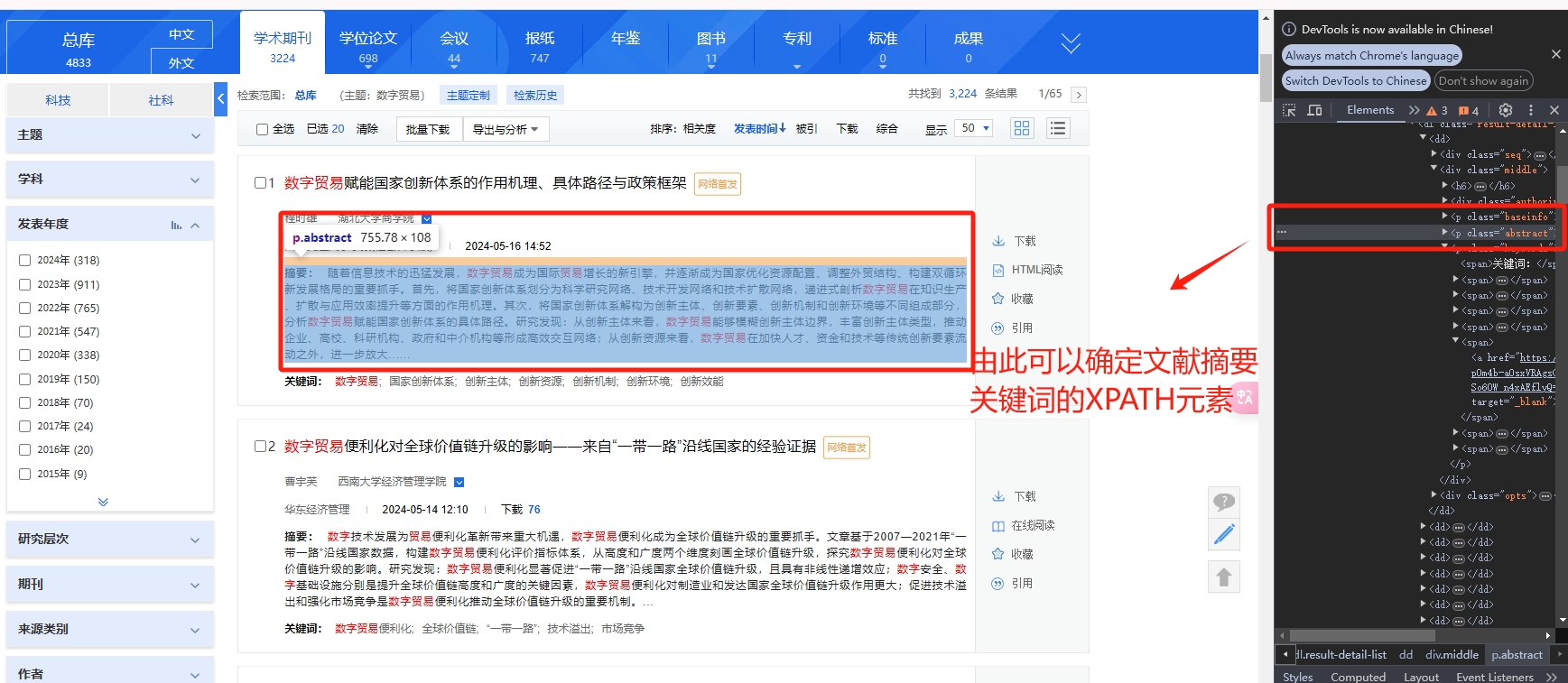
在检索完成后,应当会呈现出如下界面。在该界面中,我们能够看到符合筛选条件的文章名、发表时间、关键词、第一作者、第一作者所在机构等详细信息。我们可以通过检查页面上的元素来确定XPATH路径,进而完成页面相关元素的抓取。
下面,我们通过python来抓取这些元素。
我们拟分三批抓取论文信息
- 首先抓取详细信息,包括文章的
题目,第一作者,单位,期刊,关键词,在爬取时应当在详细页面(如上图第⑥步所示) - 其次抓取简要信息,包括文章的
题目,全部作者,期刊名称,发表时间,被引次数,下载次数,在爬取时应不用点详细模式(不用点击上图第⑥步) - 最后抓取文章的
题目和摘要
这里要说明的是,我分三次抓取的最主要原因是,知网详细界面和简略界面所显示的内容有所差异,为了全方位获取文章的信息,我们分批次抓取,最后可根据文章题目按需横向合并(merge)
下面是代码实现过程
之所以关注文章的摘要,主要是由两方面的考虑,一是便于分析文章是如何做的,用了什么方法,得到了什么结论。认知这些信息有助于我们更好地对这一支文献有整体上的把握。另外,我后续也想通过 python爬虫 + ChatGPT 来尝试生成文献综述。
在完成爬虫后,我们merge“基本信息.csv”“页面元素.csv”两个表单,进而开展相关的分析,由于两边都有文章题目,因此我们直接以题目作为match-by-key就可以。实现代码
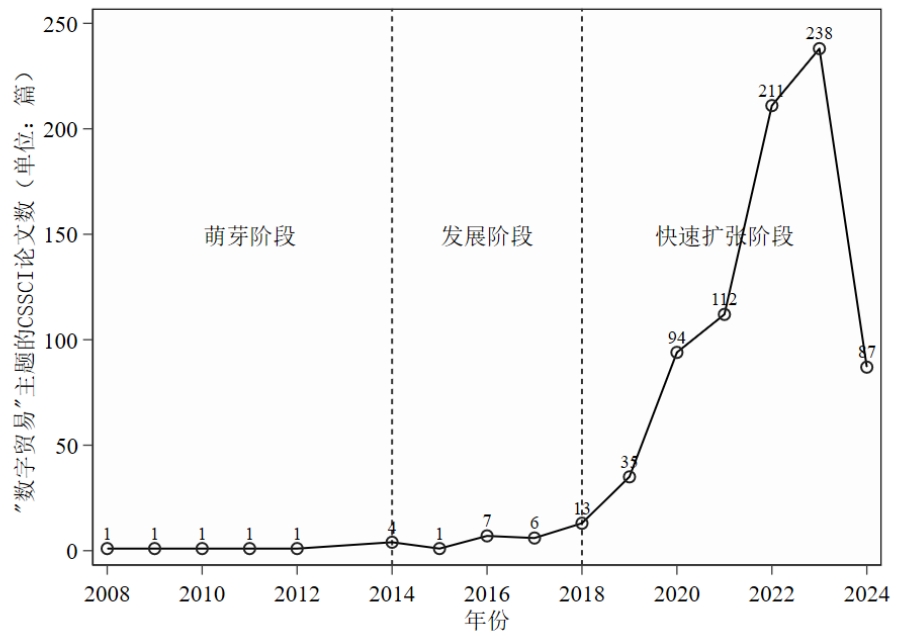
通过爬虫,我抓取了“数字贸易”相关的CSSCI文献。
我通过R语言,先对爬取后的结果进行了预处理。
个人认为R语言在处理爬取后的数据时,比python还是好一些。(作为一名经济学博士研究生,我强推R语言,它处理数据速度比stata更快,dplyr编程语法非常简单有序,而且R语言可视化难度较低,比如在这里我要画的词云图就可以通过wordcloud2宝来实现,不像python还要定义词典、去停用词等;R语言直接用R的内核+Rstudio即可完成配置,环境搭建比python容易一些,适合新手上手)
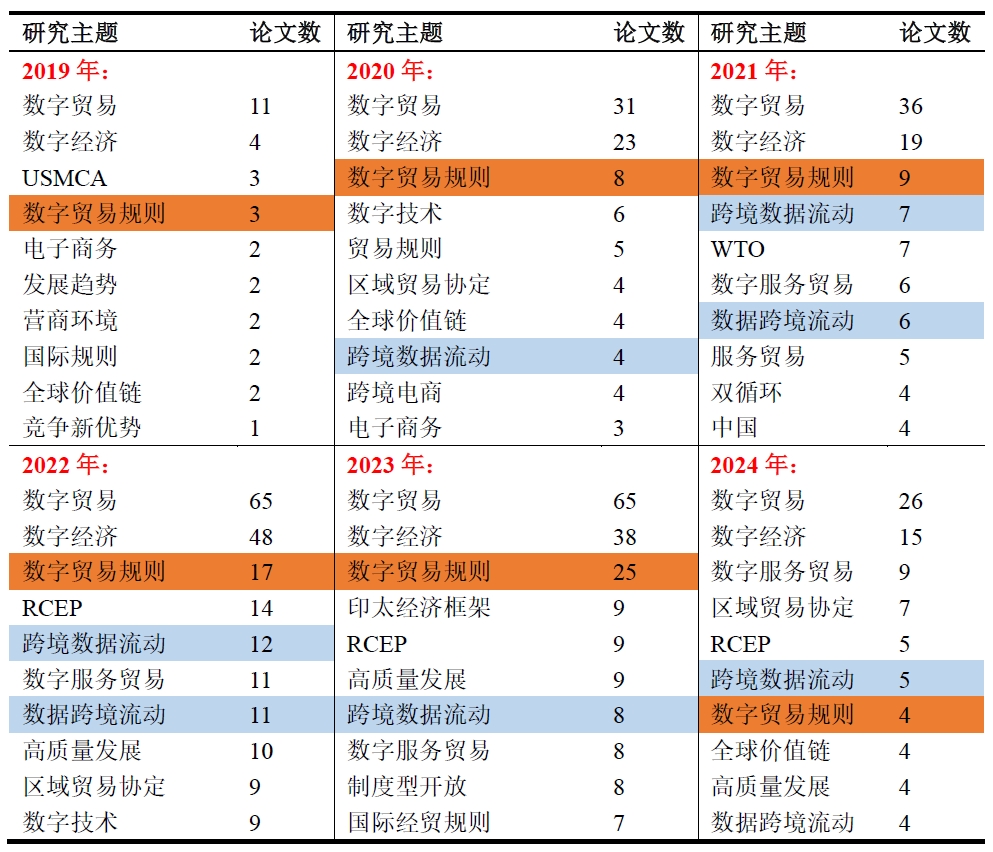
在本案例中,我以各年研究关键词的词频为dataframe,绘制了词云图。该图主要反映了各年相关研究的主题是什么。对该图进行考察有助于我们追踪相关研究的热点与研究演变历程。实现代码
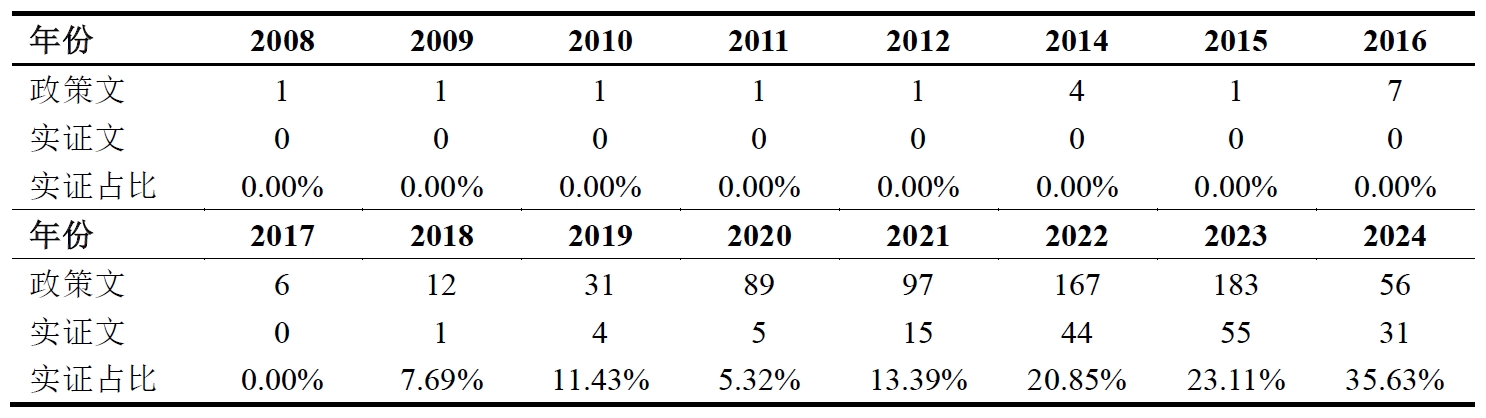
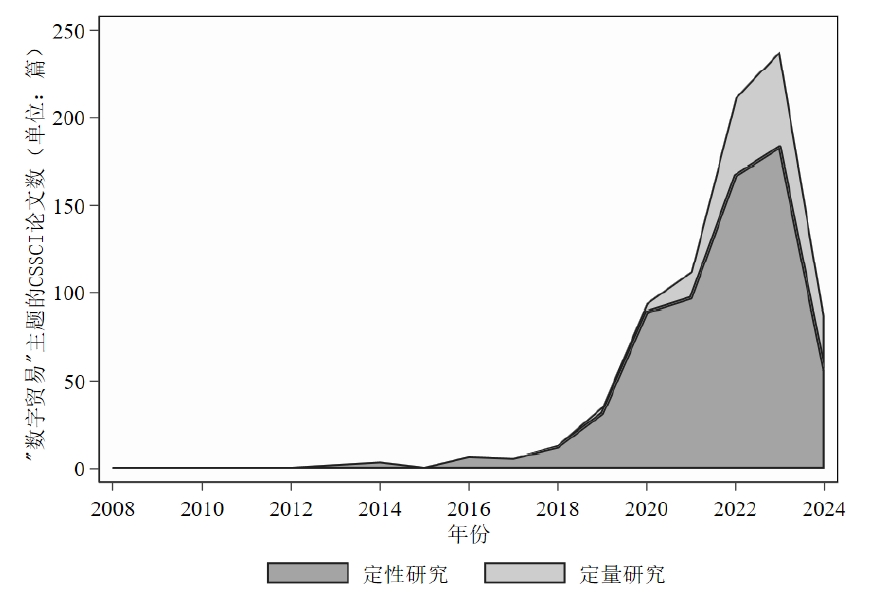
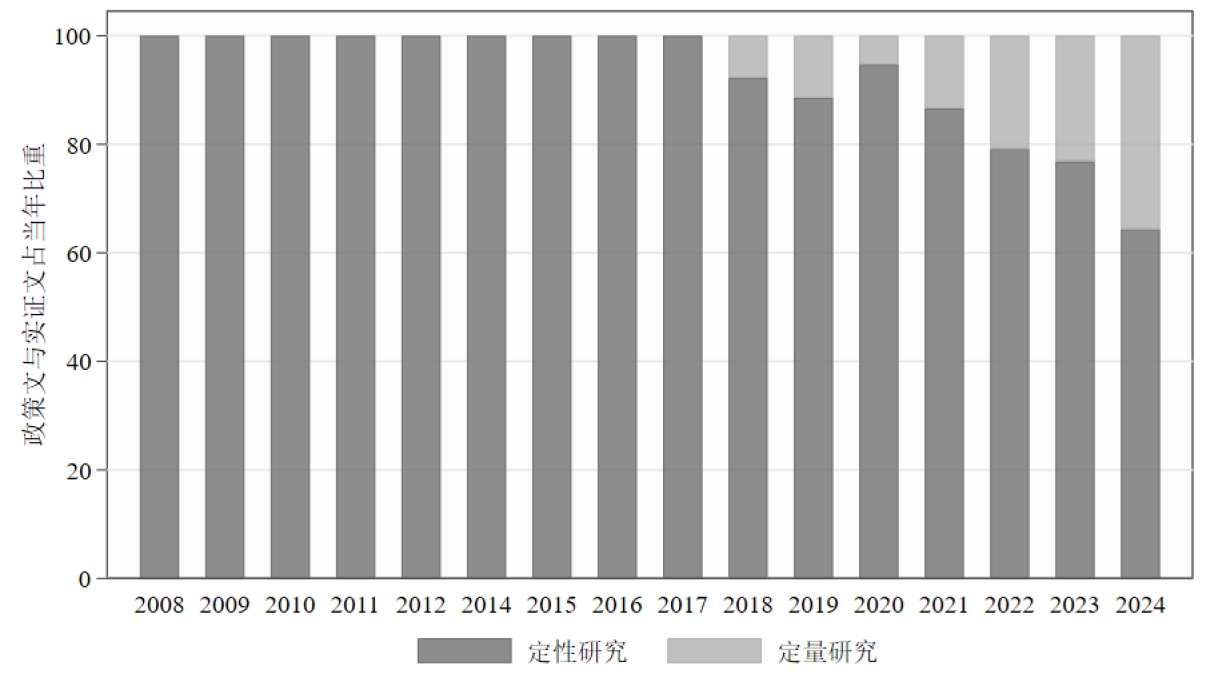
除此之外,我还基于爬下来的文章摘要,识别文章是否为定量研究。具体而言,倘若摘要中包括“实证”“数据库”“回归”“定量”这四个关键词,我就把他们定义为“定量研究”,并生成了一个新变量empirical令其等于1,否则为0。
keywords <- "实证|数据库|回归|定量"
data2<- read.csv(paste0(rootpath,"\\摘要.csv"), header = FALSE)
temp <- data2 %>%
rename(title=V1, abstract = V2) %>%
mutate(empirical= ifelse(grepl(keywords, abstract), 1, 0)) %>%
select(-abstract)
write_dta(temp,paste0(rootpath,"\\is_empirical.dta"))
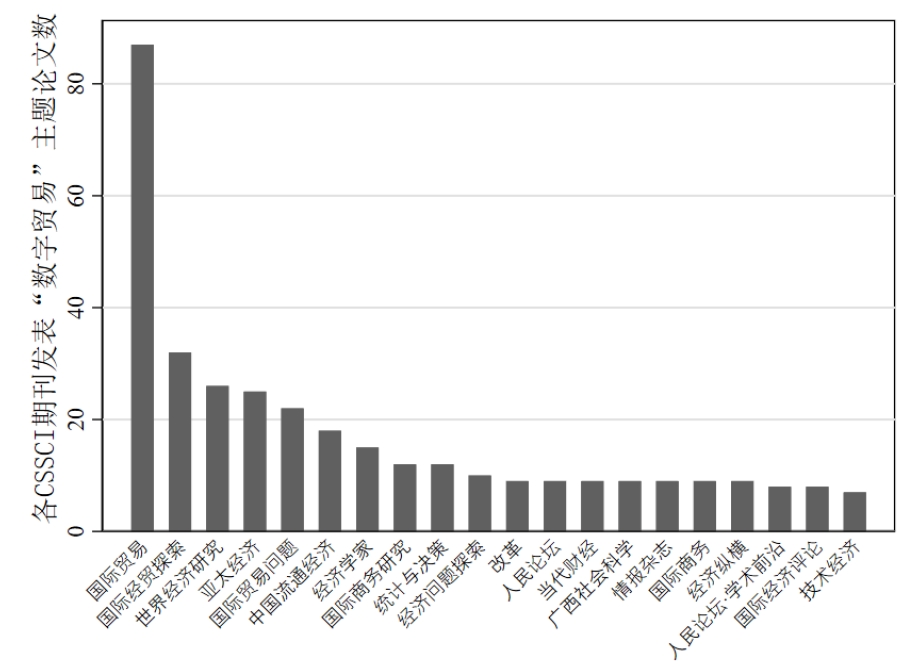
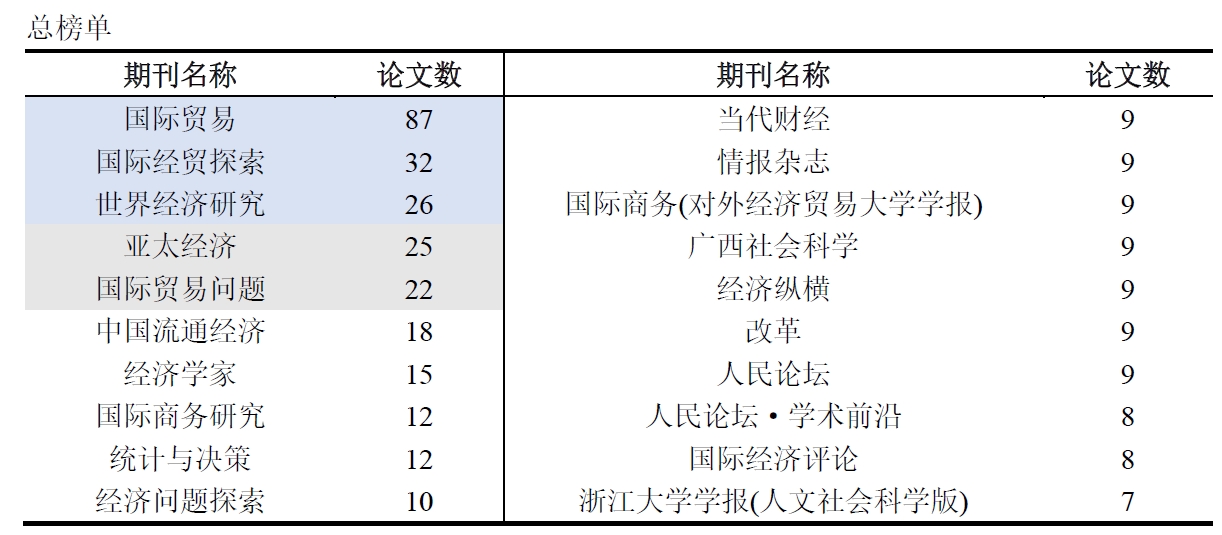
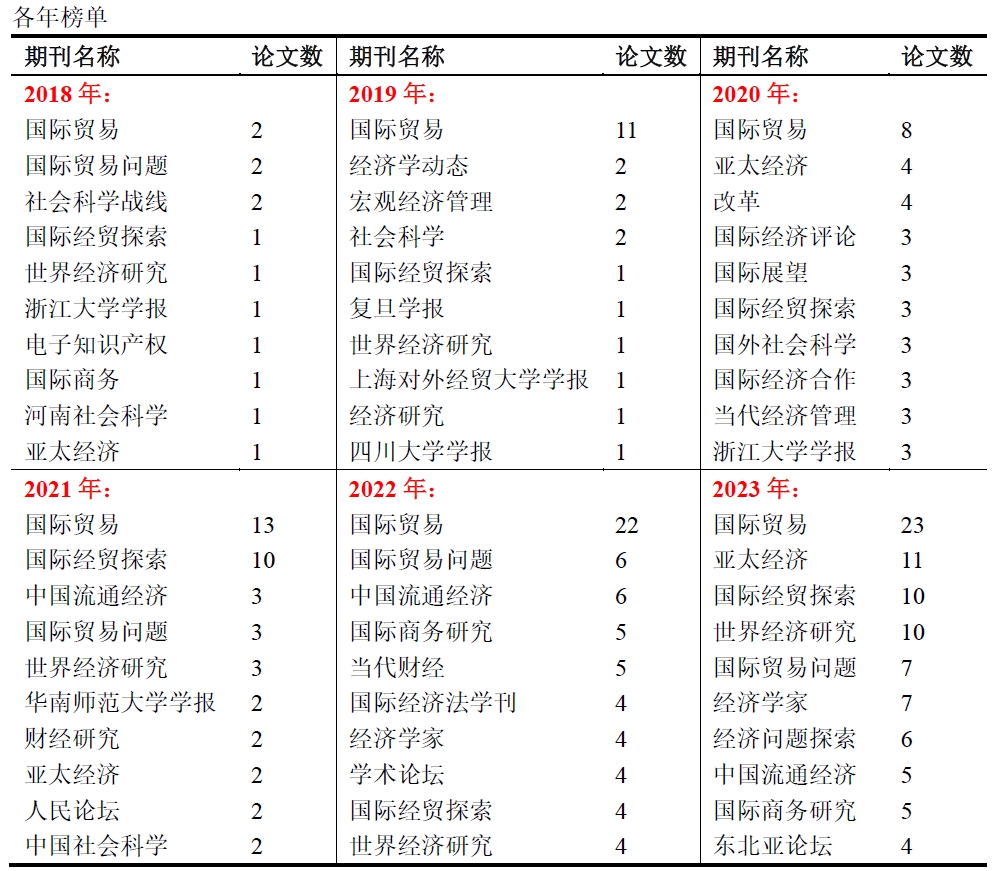
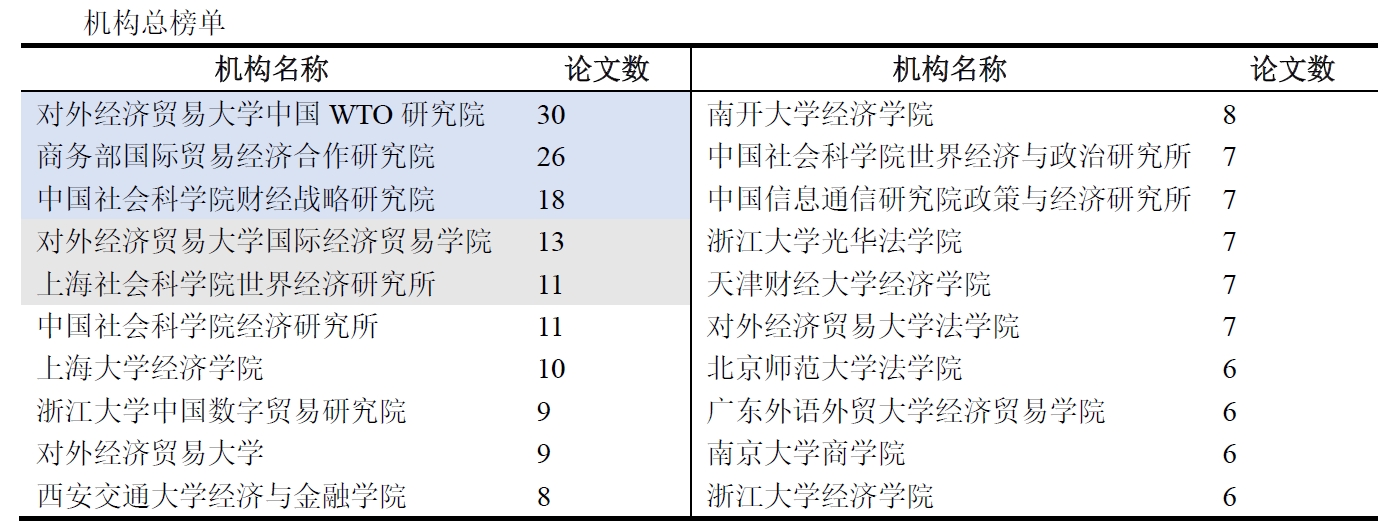
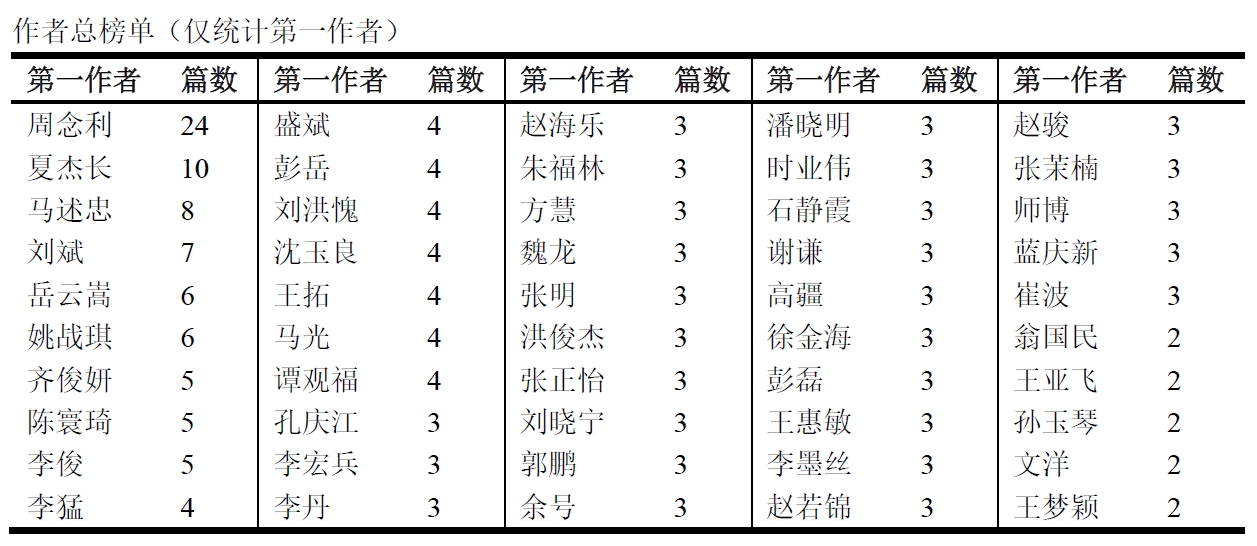
最后,我从期刊来源,作者,机构,文献类型等多个方面对爬取后的数据进行了分析,这些代码就很简单了,在此就不再解释了。直接附代码:实现代码
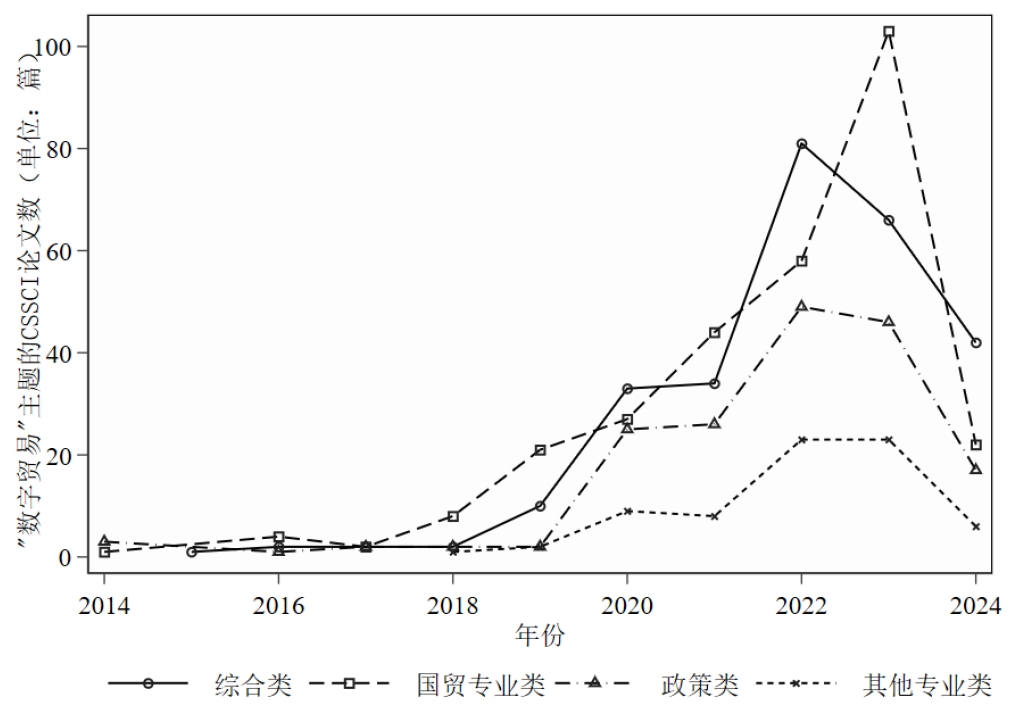
展示一下分析结果:(这些信息没什么好打码的,都是公开渠道可获的)